S-Adenosyl-L-Methionin (Ademetionin) und Epigenetik – 3.Teil: DNA-Methylierung und andere epigenetische Werkzeuge
PER | SPEKTIVE | EPI | GENE | TIK
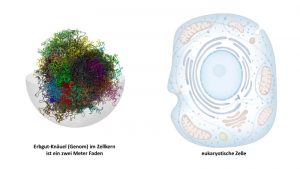
Im realen Leben sind Genetik und Epigenetik für das Lebewesen in gleicher Weise fundamental. Sie sind vollkommen komplementär zueinander und bedingen sich gegenseitig. Das Epigenom braucht das Genom, um zu funktionieren, und umgekehrt. Die epigenetischen Faktoren und Mechanismen ermöglichen die Aktivierung oder Deaktivierung von Genen und entscheiden darüber, ob sie exprimiert werden oder nicht. Es gibt also keinen Gegensatz zwischen DNA-Methylierung und andere epigenetische Werkzeuge, sondern eine stetige vitale Abhängigkeit voneinander.
Den Unterschied zwischen der Genetik und der Epigenetik kann man wahrscheinlich mit dem Unterschied zwischen dem Schreiben und dem Lesen eines Buches vergleichen. Nachdem das Buch geschrieben ist, ist der Text (die Gene oder die in der DNA gespeicherte Information) in allen an den interessierten Leserkreis verteilten Kopien der gleiche. Jedoch wird jeder Leser des Buches die Geschichte auf unterschiedliche Weise interpretieren. Das heißt, die Vorlage- das Genom ermöglicht verschiedene Lesarten – Epigenetik (Thomas Jenuwein, Freiburg).
Epigenetik
Die Epigenetik ist ein Zweig der Genetik, der sich auf die vererbbaren Veränderungen von DNA oder assoziierten Proteinen konzentriert, mit Ausnahme von Variationen der DNA-Sequenz, die während der Zellteilung einen Informationsgehalt aufweisen.
Epigenetische Veränderungen (Modifikationen)| epigenetische Werkzeuge (Mechanismen)
Das epigenetische Muster einer Zelle ist nicht starr, sondern verändert sich im Laufe der Zeit. Epigenetische Werkzeuge regulieren, in welchem Maße Gene in einer Zelle abgelesen und die gespeicherten Informationen in Proteine und Enzyme übersetzt werden. Beispielsweise können DNA-Abschnitte direkt durch chemische Gruppen (durch Methylierung, Acetylierung, Ubiquitinierung und Phosphorylierung sowie andere) markiert werden. Das hat zur Folge, dass Gene in der näheren Umgebung weniger aktiv sind. Gewöhnlich DNA-Methylierung induziert eine verminderte Protein-DNA-Bindung von Transkriptionsfaktoren und führt zur Unterdrückung der Genexpression.
Bemerkenswerterweise sind diese epigenetischen Modifikationen sowohl an der Transkriptionsaktivierung als auch an der Repression beteiligt, was auf ihre weit verbreitete Rolle als Modulatoren der Genexpression in zahlreichen biologischen Prozessen hinweist.
DNA-Methylierung
Die DNA-Methylierung ist die am meisten untersuchte epigenetische Modifikation von DNA und entspricht der kovalenten Addition einer Methylgruppe (CH 3) an das Nukleotid Cytosin. DNA „methylierbare“ Sequenzen sind im gesamten menschlichen Genom nicht einheitlich, sondern auf CpG-reiche DNA-Regionen, die als CpG-Inseln (CGI) bezeichnet werden, beschränkt.
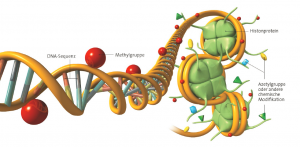
Zwischen den verschiedenen Spezies ist der Methylierungsgrad verschieden. Beim Menschen beträgt der Methylierungsgrad 1 Prozent. Es besteht keine direkte Relation zwischen der DNA-Methylierung und der Transkriptionsaktivität. Das Methylierungsprofil eines Gens ist also ein Indiz für seine Aktivität, die von anderen epigenetischen Faktoren abhängt wie den Modifikationen der mit der DNA verbundenen Histone und der nicht codierenden RNA. Die Gesamtheit dieser Faktoren wirkt zusammen, um letztlich die Aktivität eines Gens zu bestimmen.
Darüber hinaus kann die Methylierung durch einen enzymatischen Prozess der Demethylierung aufgehoben werden. Die Steuerung der Genaktivität durch Methylierung und Demethylierung der DNA ist ein extrem wichtiger und komplexer Prozess und ist in seiner Dynamik ständig unterschiedlich.
Isabelle M. Mansuy ist Professorin für Neuroepigenetik an der Medizinischen Fakultät der Universität Zürich und am Institut für Gesundheitswissenschaften und -technologie der Eidgenössischen Technischen Hochschule Zürich, in ihrem Buch: „Wir können unsere Gene steuern! Die Chancen der Epigenetik für ein gesundes und glückliches Leben“, Seite 88, Berlin Verlag 2020.
Histonmodifikationen
Die zweite Gruppe epigenetischer Veränderungen wird durch posttranslationale Histonmodifikationen (PTM) dargestellt, die in der Addition chemischer Gruppen an Aminosäurereste sowohl kanonischer Histone (H2A, H2B, H3 und H4) als auch varianter Histone (wie H3) bestehen.
Anders als bei DNA-Modifikationen gibt es mindestens acht verschiedene Arten von posttranslationalen Histonmodifikationen: Acetylierung, Methylierung, Phosphorylierung, Ubiquitylierung, Sumoylierung, ADP-Ribosylierung, Deiminierung und Prolinisomerisierung.
Im Gegensatz zur DNA-Methylierung weisen Histon-PTMs zahlreiche funktionelle Rollen auf. Beispielsweise reguliert die Histonacetylierung die DNA-Replikation, -Reparatur und -Kondensation. Methylierung, Phosphorylierung und Ubiquitylierung sind an der Reparatur oder Kondensation von DNA beteiligt.
Die Fähigkeit von Transkriptionsfaktoren, auf dieser Ebene auf nukleosomengebundene DNA zuzugreifen, wird weitgehend durch die Verpackung des Chromatins und damit der DNA gesteuert.
Es ist allgemein anerkannt, dass für ein bestimmtes Chromatinsegment eine direkte Korrelation zwischen der Histonacetylierung und der Transkriptionsaktivität besteht. Histonacetyltransferasen (HATs) erleichtern die Histonacetylierung und wirken daher vermutlich als Transkriptionsaktivatoren.
Im Gegensatz, Histondeacetylasen (HDACs) entfernen Acetylgruppen aus Histonen und unterdrücken dadurch die Transkription durch Verdichtung der DNA. Das Gleichgewicht zwischen der Aktivität von HATs und HDACs reguliert die Transkription, aber mit zunehmendem Alter geht dieses Gleichgewicht verloren, was entweder zu einer Überaktivität der Gene (z. B. in Krebszellen) oder zu einer Genrepression führt, die während des Alterungsprozesses zu einer Neurodegeneration führen kann.
Proteine der Polycomb-Gruppen
Die Proteine der Polycomb-Gruppen sind wichtige Entwicklungsregulatoren, die die Expression von Hunderten von Genen steuern. Ihre Aktivität ist notwendig, um ein „epigenetisches Gedächtnis“ spezifischer Genexpressionsmuster zu erhalten.
PcG-Proteine bilden zwei Hauptkomplexe: Polycomb-repressive Komplexe 1 und 2 (PRC1, PRC2).
Polycomb-vermittelte Gen-Stummschaltung ist bei vielen Prozessen wie der Inaktivierung und Prägung von X-Chromosomen bei Säugetieren erforderlich.
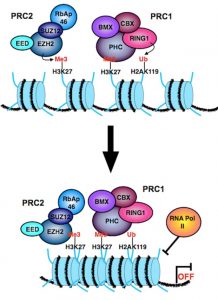
Abb. Epigenetische Gen-Stummschaltung PcG-vermittelt. PRC2 induziert EZH2-vermitteltes H3K27me3. H3K27me3 rekrutiert PRC1, das H2AK119 ubiquityliert und die Chromatin-Verdichtung und das Gen-Silencing fördert. EZH2, die katalytische Untereinheit des repressiven Polycomb-Komplexes 2, katalysiert die Addition von Methylgruppen an Lysin 27 des N-Schwanzes von Histon H3 (H3K27me). Die funktionelle Verbindung zwischen PcG-Proteinen, HDACs und DMTs zeigte eine synergistische Kontrolle der Gen-Stummschaltung, die sowohl an physiologischen als auch an pathologischen Prozessen beteiligt ist.
Referenz:
Irene Marchesi und Luigi Bagella. Rolle des Enhancers des Zeste Homolog 2 Polycomb-Proteins und seine Bedeutung für die Tumorprogression und Zelldifferenzierung. 2013. DOI: 10.5772 / 55370
Mikro-RNA – kleine Ribonukleinsäure-Moleküle
Sie sind das dritte zentrale epigenetische Schaltersystem basiert auf der Produktion von kleinen RNA-Molekülen (Mikro-RNAs), die sogenannte nicht kodierende RNAs.
Die Codes der Mikro-RNAs verstecken sich in DNA-Abschnitten, die man früher gleichermaßen für sinnlos hielt und deshalb als Müll-DNA bezeichnete.
Für die Entdeckung dieser RNA-Interferenz erhielten Andrew Fire und Craig Mello (beide USA) im Jahr 2006 den Medizin-Nobelpreis.
MicroRNAs sind kurze RNA-Moleküle, die im Gegensatz zu gewöhnlichen RNA-Strängen nicht in Proteine übersetzt werden. Ihre Funktion besteht darin, sich an andere RNA-Moleküle anzulagern und dadurch deren Übersetzung in Proteine zu verhindern oder sogar den Abbau der RNA-Moleküle zu bewirken.
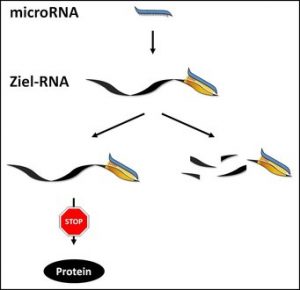
Abb. Eine microRNA lagert sich an die Ziel-RNA an und verhindert dadurch deren Übersetzung in ein Protein (links) oder löst deren Abbau aus (rechts). © MPI für Psychiatrie / Engel
Referenz:
Kim, V. N.; Han, J.; Siomi, M. C. Biogenesis of small RNAs in animals. Nature Reviews Molecular Cell Biology 10(2), 126-139 (2009)
Von einem Gen, das für eine microRNA kodiert, wird im Zellkern ein langer RNA-Strang abgelesen. Dieses Primärtranskript lagert sich zu einer Schleife zusammen und bildet dabei doppelsträngige Teilbereiche aus. Bestimmte Enzyme schneiden in einem weiteren Schritt die freien Enden des Primärtranskripts ab, so dass eine verhältnismäßig stabile, doppelsträngige Vorläufer-microRNA mit einer Länge von etwa 70 bis 110 Basenpaaren entsteht. Die Vorläufer-microRNA kann nun aus dem Zellkern in den Zellkörper transportiert werden. Dort schneidet wiederum ein spezialisiertes Enzym ein nur etwa 20 bis 22 Basen langes Stück aus dem Doppelstrang heraus. Diese fertige microRNA kann sich nun an die komplementäre Zielsequenz auf anderen RNA-Molekülen anlagern.
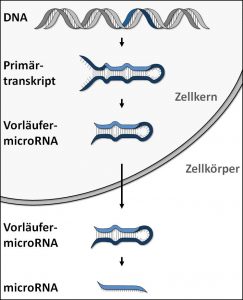
Abb.: Von einem microRNA-Gen auf der DNA wird im Zellkern ein langes Primärtranskript abgelesen, das sich zu einer Schleife zusammenlagert. Enzyme entfernen die überhängenden Enden und die entstandene Vorläufer-microRNA wird in den Zellkörper transportiert. Dort schneidet ein weiteres Enzym die reife, etwa 20 bis 22 Basen lange microRNA heraus. © MPI für Psychiatrie / Engel
Die microRNA muss nur in sechs bis acht Nukleotiden mit der Zielsequenz tatsächlich übereinstimmen. Dadurch kann sich eine microRNA oft an viele verschiedene proteinkodierende RNA-Moleküle anlagern und so die Aktivität vieler Gene gleichzeitig regulieren. Beim Menschen scheinen über 60 Prozent aller Gene durch solche microRNAs beeinflusst zu werden.
Referenz:
Friedman, R. C.; Farh, K. K.; Burge, C. B.; Bartel, D. P. Most mammalian mRNAs are conserved targets of microRNAs. Genome Research 19(1), 92-105 (2009)
Jüngste Entwicklungen haben die Existenz von Langlebigkeits-microRNAs nahegelegt, da diese nichtkodierenden RNAs die DNA-Reparatur, die oxidative Abwehr und andere Prozesse steuern, die wahrscheinlich für die Bestimmung der Lebensdauer von Individuen wichtig sind. Die Verlangsamung oder Verringerung der Dysregulation von microRNAs in der Lebensmitte kann einen wichtigen Einfluss auf die molekularen Degenerationsprozesse haben, die sonst mit dem Altern zunehmen.
Von der DNA zum Protein: Etappen der Genexpression
Die DNA-Sequenzen der Gene beinhalten alle Informationen, welche die Bildung der Proteine, als „Bausteine des Lebens“, ermöglichen.
Anfang der 50er-Jahre machten James Watson und Francis Crick eine der größten Entdeckungen des 20. Jahrhunderts. Watson, ein hochbegabter und sehr ehrgeiziger amerikanischer Biologe von gerade mal 23 Jahren, und der zwölf Jahre ältere Brite Francis Crick, ein Physiker und Biologe, wollten die Struktur der Desoxyribonukleinsäure ergründen, der DNS oder englisch DNA, die der „Baustein des Lebens aller Organismen“ ist.

Ende Februar 1953 hatten Watson und Crick schließlich jenes Modell gebaut, das heute jeder kennt: eine „Doppelhelix“. Die DNA sieht aus wie eine spiralförmig gedrehte Strickleiter, Zucker- und Phosphatmoleküle bilden die Seitenstränge und die paarweise angeordneten Basen die Stufen dazwischen.
Diese Struktur und die damit verbundene genaue Funktion der DNA beschrieben Watson und Crick in einem Bericht an die renommierte Wissenschaftszeitschrift „Nature“, die ihn am 25. April 1953 veröffentlichte. Der Artikel beginnt mit dem Satz:
„Wir möchten eine Struktur für das Salz der Desoxyribonukleinsäure – DNA – vorschlagen. Diese Struktur hat neue Eigenschaften, die von beträchtlichem biologischem Interesse sind.“
Referenz:
J. D. Watson, F. H. C. Crick: Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid. In: Nature. Band 171, S. 737–738, 25. April 1953, doi:10.1038/171737a0.
Eine einzige DIN-A4-Seite für eine der wichtigsten wissenschaftlichen Entdeckungen des 20. Jahrhunderts: Denn das Doppelhelix-Modell der DNA erklärt das Prinzip der Zellteilung, also wie sich Organismen entwickeln; und es eröffnete den Weg für ungeahnte gentechnische Anwendungsmöglichkeiten. 1962 erhielten James Watson und Francis Crick zusammen mit Maurice Wilkens dafür den Medizin-Nobelpreis.
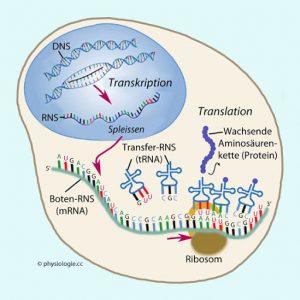
Erster Schritt: die Transkription
Der erste Schritt besteht darin, aus der DNA-Sequenz eines Gens, ob codierend oder nicht codierend, RNA zu synthetisieren. Diese Synthese vollzieht sich im Zellkern. Von Boten-RNA oder mRNA (messenger RNA) spricht man, wenn diese von einem codierenden Gen stammt, weil die DNA in eine konforme Kopie transkribiert wird, welche für die Bildung eines Proteins nötige Informationen wie ein Bote weitergibt.
Zweiter Schritt: die Translation
Der zweite Schritt besteht darin, dass die mRNA gelesen wird, um das Protein zu bilden. Nach dem „Umschreiben“ der DNA-in den mRNA-Code wird dieser nun in eine Sequenz von aufeinanderfolgenden Aminosäuren „übersetzt“. Diese Übersetzung (Translation) der mRNA in Protein vollzieht sich mithilfe der Ribosomen, die die Fähigkeit haben, die in der mRNA enthaltene Information nach dem „Codon-Prinzip“zu decodieren. Jedes Basentriplett entspricht einer bestimmten Aminosäure. Jede Aminosäure wird durch eine Transfer-RNA (tRNA) vermittelt und wird so an die vorhergehenden Aminosäuren angehängt, wie dies von der mRNA abgelesen wird. Alle Aminosäuren hängen sich so eine nach der anderen, um ein dreidimensionales Protein zu bilden.
Genexpression oder stummes Gen
Genexpression ist der Schlüsselmechanismus.
Die in unserem Genom vorkommenden Gene sind nicht alle gleichzeitig und nicht in denselben Zellen aktiv. Die epigenetischen Faktoren sind es dann, die sich darauf auswirken, ob das Genom aktiv oder inaktiv ist, ob Gene exprimiert werden oder nicht. Die epigenetischen Faktoren unterliegen wiederum selbst dem Einfluss der Umweltfaktoren.
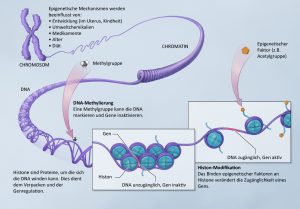
Chromatin ist das genetische Grundmaterial in unseren Zellkernen, aus dem die Chromosomen bestehen. Es handelt sich um einen Komplex aus DNA und speziellen Proteinen, von denen wiederum etwa die Hälfte Histone sind.
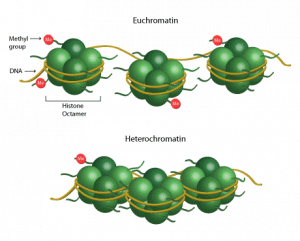
Es gibt zwei unterschiedliche Zustände der DNA: Kondensation oder Dekondensation.
Im lockeren oder dekondensierten Zustand ist das Chromatin offen; man spricht von „Euchromatin“.
Im verdichteten oder kondensierten Zustand ist das Chromatin geschlossen; hier spricht man von „Heterochromatin“.
Dieser Kondensationszustand bestimmt, ob ein Gen aktiv oder stumm ist. Ist die DNA eines Gens dekondensiert oder offen, ist der Maschinerie, die sie in RNA transkribieren kann, zugänglich. In kondensiertem, geschlossenem Zustand hingegen ist sie dieser Maschine kaum oder gar nicht zugänglich. Sie wird daher nur ansatzweise oder überhaupt nicht transkribiert werden.
S-Adenosyl-L-Methionin (Ademetionin)-Substitution



Qualitätskriterien
Die Produkte der Angewandten Epigenetik von NUGENIS enthalten S-Adenosyl-L-Methionin (Ademetionin) in präventiver Dosis, um eine DNA-Hypomethylierung vorzubeugen und damit die sich ergebenden schwerwiegenden Schäden für den Organismus zu verhindern.
Die ausgewogene Zugabe von Vitamin B12, Vitamin B6 und Folsäure mindert die Gefahr einer Hyperhomocysteinämie.
Durch den hohen (S,S)–S-Adenosyl-L-Methionin (Ademetionin)-Anteil = aktive Form des S-Adenosyl-L-Methionin (Ademetionin) wird seine biologische Wirkung garantiert.
S-Adenosyl-L-Methionin (Ademetionin) wird bei der Einnahme vor dem Zugriff der Magensäure geschützt. Erstens durch eine magensaft-resistente Kapsel und zweitens als säurefestes gecoatetes S-Adenosyl-L-Methionin (Ademetionin)-Granulat.
GESUND SEIN UND GESUND BLEIBEN
S-Adenosyl-L-Methionin (Ademetionin) und Epigenetik –
– 1.Teil: Die metabolische Vielfältigkeit von S-Adenosylmethionin (Ademetionin)
– 2.Teil: Die mitochondriale Dysfunktion bei S-Adenosyl-L-Methionin (Ademetionin) – Mangel
– 4.Teil: Die durch S-Adenosyl-L-Methionin (Ademetionin) – Mangel induzierte DNA-Untermethylierung und die sogenannten „Volkskrankheiten“
Ihr Eduard Rappold
