Herz – Kreislauf – Gesundheit: Epigenetik – Genmethylierung, Transkriptionsfaktoren und nicht kodierende RNAs
Herz – Kreislauf – Gesundheit:
Epigenetik – Genmethylierung, Transkriptionsfaktoren und nicht kodierende RNAs
Epigenetische Mechanismen steuern die Entwicklung, Reifung und Funktion von Herzmuskelzellen
Methylierungen beeinflussen die Genaktivität
Drei epigenetische Mechanismen werden im Wesentlichen unterschieden: DNA-Methylierungen, Histon-Modifikationen und nicht kodierende RNAs.
Eine wichtige Rolle spielen beim epigenetischen Prozess Methylgruppen, die von Enzymen (DNA-Methyltransferasen, DNMTs) an bestimmten Stellen der Erbsubstanz (DNA, Histone) angeheftet werden. Gemeinhin führt diese DNA/Histon-Methylierung zu einer Inaktivierung des betreffenden Gens.
DNA-Methylierungen gelten als die stabilste, am wenigsten dynamische epigenetische Modifikation. Durch den gegensätzlichen Prozess, der Demethylierung, können Zellen Methylierungen über einen komplexen Stoffwechselweg wieder entfernen.
Methylierung der DNA per se: In der DNA sind die Nukleinbasen Adenin, Thymin, Guanin und Cytosin nicht gleichmäßig verteilt. Größere „CpG-Inseln“ enthalten vermehrt Cytosin und Guanin direkt nebeneinander, in diesen Bereichen können Methylierungen am Cytosinmolekül stattfinden.
DNA-Hydroxymethylierung Ähnlich wie bei der DNA-Methylierung ist die DNA-Hydroxymethylierung ein epigenetischer Mechanismus, der die 5-Position von Cytosin durch Hinzufügen einer Hydroxymethylgruppe zur DNA modifiziert. Obwohl dieser Mechanismus nicht vollständig verstanden ist, wird angenommen, dass die DNA-Hydroxymethylierung an zahlreichen wichtigen biologischen Prozessen einschließlich der Differenzierung beteiligt ist und die Genexpression beeinflussen kann.
Die Verteilung von 5-hmC ist in menschlichen Geweben gewebeabhängig und der Gehalt an 5-hmC variiert in Krebszellen.
Die Umwandlung von Cytosin (C) in 5-Methylcytosin (5-mc) erfolgt durch DNA-Methyltransferase-Enzyme (DNMTs) und in 5-Hydroxymethylcytosin (5-hmC) durch TETs (zehn elf Translokation).
DNA-Methylierungstäler sind Regionen, die weitgehend davor geschützt sind, durch Anlagerung von Methylgruppen in den Modus der Nichtaktivierbarkeit versetzt zu werden. Hier gesteuerte Gene sind offenbar so wichtig, dass sie jederzeit schnell und mit perfektem Timing von Transkriptionsfaktoren an- und ausgeschaltet werden müssen.
Transskription und Proteinsynthese
https://www.youtube.com/watch?v=Fy_3gpIkoNs
https://www.youtube.com/watch?v=HSdK7l9Qk1w
https://www.youtube.com/watch?v=3wFfj6D0_nQ
Transkriptionsfaktoren bestimmen die Transkriptionsrate
Transkriptionsfaktoren sind Proteine, die die Umsetzung der DNA in RNA steuern und so dafür sorgen, dass ein Protein in der jeweils benötigten Menge gebildet wird. Sie binden an bestimmte DNA-Abschnitte und regulieren, wie viel Boten-RNA (mRNA) davon hergestellt wird.
Spezifische Transkriptionsfaktoren werden meistens durch Proteinkinasen, Enzyme, die den Transfer einer Phosphatgruppe von einem Donor (meist ATP) auf die Seitenketten-Hydroxyl-(OH-)Gruppe einer Aminosäure katalysieren, aktiviert.
NF-AT (nuclear factor of activated T-cells) ist für die Genexpression von Zytokinen verantwortlich. NF-κB (Nukleärer Faktor κB) hat zahlreiche Zielgene und vermittelt unterschiedlichste Transskriptionswirkungen. Tumorsuppressorprotein p53 ist an der Kontrolle des Zellzyklus, an der Induktion der Apoptose (des programmierten Zelltods) oder an der DNA-Reparatur beteiligt. STAT-Proteine (Transducers and Activators of Transcription) sind an vielen Aspekten von Zellwachstum und Proliferation beteiligt. CREB (cAMP response element-binding protein); als erster Weg zur Aktivierung von CREB wurde die Signaltransduktion über cAMP und Proteinkinase A unter anderem durch den Nobelpreisträger Eric Kandel beschrieben. Myogenin ist ein Transkriptionsfaktor, der an der Aktivierung muskelspezifischer Gene beteiligt ist.
Die Aktivität von Transkriptionsfaktoren wird bestimmt durch deren Regulation.
- Sie binden den RNA-Polymerasekomplex. Dies verleiht der Polymerase eine höhere Bindungs-Affinität zu dem aktivierten Promoter, dieser wird also nun verstärkt gebunden, beziehungsweise die Promoterstärke wird erhöht und die nachfolgende proteincodierende Sequenz wird verstärkt exprimiert.
- Transkriptionsfaktoren haben Histon-Acetyl-Transferase Funktion oder rekrutieren solche. Durch die Acetylierung von Histonen wird das Chromatin aufgelockert, wodurch die RNA-Polymerase besser Zugang zur DNA bekommt. Sie kann daher besser an diese binden und damit kann auch effizienter transkribiert werden.
Repressoren von Transkriptionsfaktoren funktionieren nach einem umgekehrten Prinzip. Histon-Deacetylasen führen zu einer dichteren Verpackung der DNA und durch die Blockade von Polymerasebindestellen folgt das Absenken der Bindungs-Affinität. Eine komplexe Regulation kommt durch das netzwerkartige Zusammenspiel vieler verschiedener Transkriptionsfaktoren zustande.
Methylierung der Histone: Diese Strukturproteine, die wie Spannfedern die DNA-Helices in der gewünschten platzsparenden Form halten, haben auch Aufgaben in der Informationsverarbeitung. Sie werden vor allem an ihren N- und C-terminalen Enden methyliert, aber auch durch zahlreiche andere Modifikationen (wie Acetylierung und Phosphorylierung) in ihrer Aktivität gesteuert.
Histon-Modifikationen (-Bindungen) bestimmen den Verpackungsgrad der DNA
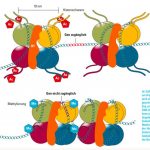
Abb. Histon-Modifikationen bestimmen den Kondensationsgrad der DNA. Im Zellkern ist die DNA um Histone gewickelt. Siewird dadurch komprimiert,geordnet und geschützt.Eng um Histone gewickelte DNA (Heterochromatin) kann jedoch nicht abgelesen werden. Azetylierungen (Ac) und Methylierungen (Me) der Histon-Seitenkettenändern diese Interaktionen. Azetylierung fördert so die Genaktivität.
Die DNA ist im Zellkern des Menschen um Histon-Proteine gewickelt. Hierdurch werden die langen DNA-Moleküle in einer geordneten Struktur soweit komprimiert, dass sie in den Zellkern passen. Aufgrund von elektrochemischen Wechselwirkungen der sauren DNA mit den basischen Histonen führt diese Bindung gleichzeitig dazu, dass die DNA nur noch bedingt für den Ableseprozess (die Transkription) zur Verfügung steht. Durch chemische Modifikation von Seitenketten der Histone kann die Stärke der Bindung jedoch modifiziert werden. So lockert z.B. die Azetylierung von Lysin an Position 27 in Histon 3 oder die Trimethylierung von Lysin an Position 4 in Histon 3 die Bindung und ermöglicht die Genexpression. Eine Vielzahl von Enzymen kann die Histone modifizieren, indem sie Modifikationen entweder erzeugen (»Writer«) oder löschen (»Eraser«). Eine dritte Klasse von Enzymen, die »Reader«-Proteine, sind darüber hinaus in der Lage, Histon-Modifikationen zu erkennen und andere Proteine an die DNA binden.
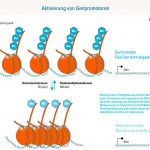
Abb. Aktivierung von Genpromotoren. Histon3-Lysin4-Trimethylierung markiert aktive Genpromotoren. Promotoren sind regulatorische Elemente von Genen, die deren Ablesung und Aktivität kontrollieren. Dreifache Methylierung von Histon3 an Lysin4 (H3K4me3) führt zur
Promoteraktivierung und markiert damit aktive Promoter. Demethylierung führt zur Promotorinaktivierung.
Histon-Modifikationen sind wesentlich dynamischer als die DNA-Methylierung.
Methylierung der RNA: Neben der bekannten Messenger-RNA gibt es zahlreiche nicht-kodierende RNA-Moleküle, die man nach ihrer Größe grob in „small non-coding RNA“ (microRNA, kurz miR) und „long non-coding RNA“ (LncRNA) einteilen kann. Auch in der RNA kann Cytosin methyliert werden.
Nicht kodierende RNAs
Es war lange Zeit rätselhaft, dass nur ca. drei Prozent der umgeschriebenen menschlichen DNA für Proteine kodieren. Der Rest der DNA galt als evolutionärer Müll (»Junk-DNA«). Diese Bereiche der DNA haben jedoch vielfältige Funktionen und sind u. a. der Ursprung von RNA- Molekülen, die rein regulatorische Funktionen haben. Unter anderem können kurze RNAs (»microRNAs« – miRs) proteinkodierende RNAs destabilisieren und so die Proteinexpression hemmen. Längere nicht kodierende RNAs (lncRNAs) werden beispielsweise benötigt, um das zweite X-Chromosom der Frau zu inaktivieren.
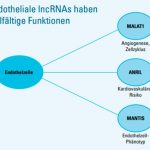
Im weiteren haben Endotheliale IncRNAs vielfältige Funktionen für Herz und Kreislauf:
Die lncRNA MALAT1
MALAT1 steht für »Metastasis Associated Lung Adenocarcinoma Transcript 1«. MALAT1 war die erste lncRNA, für die eine Bedeutung im vaskulären System gezeigt wurde. Der Verlust von MALAT1 reduziert die Angiogenese (Gefäßneubildung) und fördert die Atherosklerose.
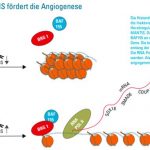
Abb. Die IncRNA fördert die Angiogenese. Die Histondemethylase JARID1b vermittelt die Inaktivierung des MANTIS Promoters. Herabregulation von JARIB1b induziert MANTIS. Diese rekrutiert darauf BRG1 und BAF155 an die DNA im Bereich angiogener Gene. Die beiden Proteine verschieben Histone entlang der DNA und öffnen so das Chromatin. Die RNA-Polymerase II kann darauf rekrutiert werden. Als Folge wird die Expression von angiogenen Genen gesteigert.
Anril – lncRNAs als kardiovaskulärer Risikofaktor?
Eine DNA-Region auf Chromosom 9 (Chr9p21) trägt einen Risikofaktor für das Auftreten von Atherosklerose. Nur befindet sich in dieser Region kein für ein Protein kodierendes Gen, vielmehr liegt dort die lncRNA ANRIL (antisense non coding RNA in the INK4 locus). Interessanterweise zeigte sich, dass der genetische Status die Produktion des lncRNA ANRIL beeinflusst. Beim Splicing werden nach der Transkription die nicht codierten Bereiche (Introns) aus dem RNA-Strang herausgeschnitten. Übrig bleiben die Exons, die zusammen mit dem gecappten und polyadenylierten RNA-Enden die gereifte mRNA bilden. Diese wird anschließend aus dem Zellkern in das Cytoplasma transportiert. Durch alternatives Splicen wird entweder ein langes lineares RNA-Molekül produziert oder eine kreisförmige, zirkuläre RNA. Zirkuläres ANRIL versetzt glatte Gefäßmuskelzellen und Fresszellen der angeborenen Immunität (Makrophagen) in den Ruhezustand. Lineares ANRIL regt sie dagegen zum Wachsen und Auswandern an, was Atherosklerose begünstigt.
MANTIS schützt vor Atherosklerose
Da Endothelzellen das Innere von Blutgefäßen auskleiden, sind sie ständig dem Blutstrom ausgesetzt. Dieser erzeugt an den Zellen eine Schubspannung, die einen schützenden Effekt auf die Zellen hat und die endotheliale Genexpression beeinflusst. MANTIS ist eine der lncRNAs, die in dieser Situation von der Zelle produziert wird. Funktionell rekrutiert MANTIS einen Chromatin-Remodelling-Komplex namens SWI/SNF (SWItch/Sucrose Non-Fermentable), so dass die Histondekoration der DNA verändert wird. Infolgedessen führen Endothelzellen ein spezielles genetisches Programm aus, was zur Ausprägung ihres endothelialen Phänotyps führt. MANTIS ermöglicht z.B., dass sich Endothelzellen entlang des Blutstroms ausrichten und befähigt sie zur Gefäßneubildung.
Quelle: Prof. Dr. Ralf Brandes https://www.forschung-frankfurt.uni-frankfurt.de/83884237.pdf
Epigenetische Regulation von Herz-Kreislauf-Erkrankungen
Da die Epigenetik starke Einflüsse auf die Genexpression hat, ist es nicht überraschend, dass sie bei chronisch verlaufenden Herz-Kreislauf-Erkrankungen eine wichtige Rolle spielt.
So wirken Hemmstoffe der Histon-modifizierende Enzyme aus der Familie der HDACs (Histondeacetylaseinhibitoren) hemmend auf das überschießende Wachstum glatter Muskelzellen nach Gefäßverletzung z.B. im Rahmen einer Ballondilatation eines verengten Herzkranzgefäßes.
Nach einem Herzinfarkt kommt es häufig zu ungünstigen Umbauprozessen des verbleibenden Herzgewebes (»Remodelling«), was ebenfalls durch HDAC-Inhibitoren gehemmt werden kann.
Schließlich fördern die Substanzen die Bildung neuer Blutgefäße (Angiogenese). Die Kombination von verstärkter Gefäßneubildung bei gleichzeitigem Schutz des Herzens machen HDAC-Inhibitoren zu attraktiven Molekülen in der Therapie von Herz-Kreislauf-Erkrankungen. ,
JQ1 ein Bromodomain-Inhibitor, hemmt BRD4 (Gen) mit dem Chromatin-Reader-Protein, das acetylierte Histone erkennt und bindet und eine Schlüsselrolle bei der Übertragung des epigenetischen Gedächtnisses über Zellteilungen und Transkriptionsregulation spielt und reduziert z.B. ebenfalls das ungünstige Remodelling nach Herzinfarkt. Darüber hinaus induziert JQ1 als Autophagie-Induktor die Akkumulation von Autophagosomen und Autolysosomen sowie den Autophagiefluss.
Von menschlichen Genom können weit mehr als 10.000 nicht kodierende RNAMoleküle abgeschrieben werden. Somit ist es möglich, nicht kodierende RNAs als Marker zu identifizieren, die z.B. nur im Erkrankungsfall produziert werden, nur in der erkrankten Zelle vorliegen und spezifisch die Erkrankung vermitteln. Am besten sind bisher die microRNAs untersucht: Diese können im Tiermodell sowohl fördernd (z.B. miR-21, miR-208) als auch hemmend (z.B. miR-133, miR-29) auf die Entwicklung der Herzinsuffizienz wirken.
Ähnliches gilt für das Gefäßwachstum, z.B. nach Verletzung durch Ballondilatation oder bei der Entwicklung einer Atherosklerose. miR-23b und miR-126 hemmen den Prozess, während miR-21, miR-29 und miR-92a ihn fördern.
Herzmuskelzellen durchlaufen einen Reifungsprozess
In Herzmuskelzellen sind etwa 10 000 Gene aktiv, die für Proteine kodieren. 5% dieser Gene sind spezifisch nur in Kardiomyozyten exprimiert. Epigenetische Regulatoren spielen eine wichtige Rolle bei der Herzentwicklung: Bei bis zu 10% der angeborenen Herzfehler konnten Genmutationen – vor allem in epigenetischen Regulatoren – festgestellt werden.
In der Entwicklungsbiologie hat Epigenetik die Aufgabe, den Entwicklungsprozess und somit die zelluläre Differenzierung in eine Richtung zu lenken. Beim ausgewachsenen Organismus fördert Epigenetik dagegen Stabilität, reduziert schädliche Umwelteinflüsse und limitiert die Schwankungsbreite der zellulären Reaktion.
Das Herz ist das erste Organ, das sich bei der Embryonalentwicklung bildet. Es muss von der frühesten Stunde in der fünften Schwangerschaftswoche bis in das hohe Alter kontinuierlich schlagen. Die vergleichsweise großen Herzmuskelzellen entwickeln sich aus Vorläuferzellen, die anfänglich, wenn das Organ sehr stark wächst, noch die Fähigkeit zur Teilung besitzen. Schon rasch nach der Geburt verlieren die meisten Herzmuskelzellen diese Fähigkeit und müssen sich auf anderem Weg an wechselnde physiologische Gegebenheiten anpassen. Sie verlieren also einen wichtigen Reparaturmechanismus, der in anderen Körpergeweben lebenslang aufrechterhalten wird. Nach einem Infarkt können nur wenige Herzzellen durch Zellteilung regenerieren, die meisten Zellen müssen sich nach einer Erkrankung anders adaptieren, um Schädigungen auszumerzen.
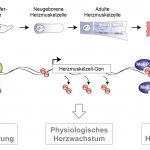
Abb. In der Entwicklung durchlaufen Herzmuskelzellen einen Reifungsprozess (oben), der von der DNA epigenetisch gesteuert wird (unten). Im Verlauf werden Methylgruppen (-CH3) von der DNA entfernt, um Transkriptionsfaktoren (TF) den Zugang und das Ablesen der Gene zu ermöglichen. Bei Herzinsuffizienz wird das pathologische Wachstum durch Proteine gesteuert, die die DNA-Methylierung erkennen (z.B. das Protein MeCP2 bindet methylierte CpG-Dinukleotide in DNA-Promotoren und führt so zur Aktivierung/Inhibierung der Transkription).
© Prof. Dr. Lutz Hein, Universität Freiburg.
Ein Herz schlägt vor der Geburt unter anderen Sauerstoff- und Nahrungsbedingungen als danach. Der Übergang vom prä- zum postnatalen Leben erfordert Anpassungen, die das Organwachstum gleichzeitig zur optimalen Kontraktionsfunktion ermöglichen. Die Energie gewinnt der Kardiomyozyt vorgeburtlich aus Glucose, später vor allem aus Fettsäuren. Daraus ergeben sich unterschiedliche Aufgaben an die Herzmuskelzelle, die sie mit der Aktivierung verschiedener epigenetischer Programme bewältigt. Ein umgestellter Stoffwechsel erzeugt andere Metaboliten und es werden andere Komponenten in den kontraktilen Zellapparat aus Aktin- und Myosinfilamenten eingebaut.
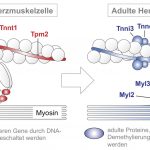
Abb. Unterschiedliche Proteinzusammensetzung der Kontraktionsfasern einer Herzmuskelzelle vor und nach der Geburt zum Zweck einer optimalen Anpassung des Herzens an die Versorgung mit Sauerstoff und Nährstoffen.
Die bis zur Geburt verwendeten Proteine (links) werden durch DNA-Methylierung ihrer Gene nach der Geburt abgeschaltet (rot). Die an diese Stelle tretenden Proteine (blau) werden nach der Geburt durch Demethylierung der DNA angeschaltet.
© Prof. Dr. Lutz Hein, Universität Freiburg.
Herzmuskelschwäche und epigenetische Vererbung
Bei Erkrankungen des Herzmuskels, wie den Kardiomyopathien, spielen neben Genmutationen auch epigenetische Mechanismen eine Rolle. Kardiomyopathien sind Erkrankungen des Herzmuskels selbst, die also nicht indirekt als Schädigung durch einen Herzinfarkt oder durch Herzklappendefekte verursacht wurden. Es besteht Gefahr einer Herzinsuffizienz bis hin zum plötzlichen Herztod.
Bei der Herzinsuffizienz mit deutlich eingeschränkter Pumpkraft findet ein enormes Größenwachstum der Zellen statt und Methylierungen in der DNA spielen eine bedeutende Rolle bei der Ausprägung dieses Krankheitshildes.
Bei dilatativer Kardiomyopathie (DCM) oder Herzversagen finden sich Methylierungsunterschiede bei vier Genen und zwar bei „Lymphocyte antigen 75“ (LY75), „Tyrosine kinase-type cell surface receptor HER3“ (ERBB3), „Homeobox B13“ (HOXB13) und „Adenosine receptor A2A“ (ADORA2A). Die Methylierung bewirkte bei den DCM-Patienten eine fast vollständige Blockierung der mRNA-Expression der beiden Gene LY75 und ADORA2A.
Gehirn –Herz – Achse
Gehirn und Herz kommunizieren unter normalen physiologischen Bedingungen ständig.
Die neuroanatomische Grundlage der „Brain-Heart“-Achse ist der Baroreflex-Bogen
In den Karotiden, im Aortenbogen, im Herzen und im pulmonalen Kreislauf befinden sich die Barorezeptoren. Diese Mechonorezeptoren registrieren fortlaufend den aktuellen Blutdruck und leiten die Information durch den Vagus und den Glossopharyngeus an den Nucleus tractus solitarii im Hirnstamm. Dieser gibt das Signal an andere autonome Kerngebiete bzw. Zentren weiter. Einerseits an den Hypothalamus und in weiterer Folge an corticale Areale, wie z.B. die Insulae, welche die unbewussten Körperfunktionen ständig an unsere Gedanken, Gefühle und an die Umwelt anpassen, andererseits aber auch an den efferenten Schenkel, welcher aus dem Nervus vagus und den prä-und postganglionären sympathischen Nervenfasern zum Herzen, den peripheren Gefäßen und den Nieren besteht.
Die epigenetischen Folgen von Herzinsuffizienz auf die Gehirnfunktion
Die Prävalenz von Depressionen und Angstzuständen liegt bei Patienten mit Herzinsuffizienz bei 21% bzw. 13–30%. Auch haben Patienten mit Herzinsuffizienz ein erhöhtes Risiko, eine Demenz zu entwickeln.
Eine durch Herzinsuffizienz induzierte kognitive Einschränkung ist mit epigenetischen Veränderungen verbunden, die die Genexpression in Neuronen des Hippocampus beeinflussen. Herzfunktionsstörungen führen zu einer Veränderung der Transkriptionslandschaft in Neuronen des Hippocampus, einer Struktur, die eine wichtige Rolle für das Gedächtnis und das Lernen spielt. Hippocampus-Neurone weisen im Verlauf der Herzinsuffizienz ein dysreguliertes Genexpressionsprofil auf und Herzinsuffizienz beeinflusst stark die Expression von oxidativen und endoplasmatischen Retikulum- Stressgenen in Hippocampus-Neuronen. Durch eine fokale zerebrale Ischämie bei verminderte Perfusion des Gehirn werden stressinduzierter Gene, (Hämoxygenase, Hitzeschockproteine, Glukose-regulierten Proteine, Glukosetransporter) exprimiert.
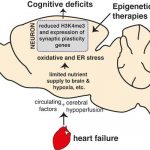
Abb. Eine verminderte Perfusion des Gehirns, die auch den Hippocampus betrifft und mit einer verminderten Herzfunktion bei Herzinsuffizienz zusammenhängt, löst Veränderungen in der Genexpression aus.
Übliche Histonmodifikationsmarkierungen befinden sich im dynamischen Gleichgewicht, und der Grad der Acetylierung / Deacetylierung und Methylierung / Demethylierung spezifischer H3K-Reste hängt von der Aktivität spezifischer Proteine ab.
Histone-lysine N-methyltransferase 2A (KMT2a), eine der beiden K4-Methylasen in Hippocampuszellen bei Herzinsuffizienz, wird weniger exprimiert. Dieser Befund verbindet die Modifikation der Genexpression mit einer verringerten H3K4me3 (Tri-methylation of lysine 4 on histone H3) -Ablagerung durch eine verringerte Expression dieser spezifischen Methylase.
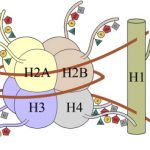
Abb. Histone kommen in Komplexen vor, die als Nukleosomen bezeichnet werden. Jedes Nukleosom besteht aus acht Histonen (normalerweise zwei Kopien von H2A, H2B, H3 und H4), die durch 147 bp DNA gebunden sind. Viele chemische Modifikationen finden sich an den Schwänzen der Histone. Diese umfassen, ohne darauf beschränkt zu sein, Acetylierung , Mono-, Di- oder Trimethylierung und Ubiquitinierung und können in verschiedenen Aminosäuren an den Schwänzen der verschiedenen Histone auftreten.
Inhibitoren von Histon-Acetyltransferasen (HAT’s) wie Spermidin wirken als potente Autophagie-Induktoren
EP300: E1A-bindendes Protein p300, eine Lysin-Acetyltransferase, N ( α )-Acetyltransferase 20, auch bekannt als NAT5, katalysiert die N-terminale Acetylierung von Methioninresten.
EP300 wirkt als endogener Repressor der Autophagie.
Als potente Autophagie-Induktoren wirken Anacardinsäure, Curcumin, Garcinol einschließlich Spermidin de facto als EP300-Inhibitoren und deacetylieren zelluläre Proteine, hemmen den mTORC1 (Säugetier-Target des Rapamycin-Komplexes 1)- Signalwegs und induzieren die Autophagie.
Zwei Arten von Enzymen, die Histon-Desacetylasen (HDAC) und Histon-Acetyltransferasen (HAT) regulieren dabei das Gleichgewicht des Acetylierungsstatus von nukleosomalen Histonen.
Anacardinsäure (isoliert aus Schalen von CashewNüssen) ist ein nichtkompetitiver Hemmer von p300 und PCAF-HAT Aktivität.
Balasubramanyam, K. et al.2003
Curcumin war der erste bekannte p300/CBP-spezifische zelldurchlässige HAT-Inhibitor. Die Kombination von Curcumin mit anderen Derivaten wie Demethoxycurcumin und Bisedethoxycurcumin bildet die Kurkuma. Curcumin aus der Gelbwurz (Curcuma longa) hemmt p300.
Marcu, M. G. et al. 2006
Garcinol (isoliert aus der Fruchtrinde von Garcinia indica) hemmt die PCAF- und p300-HAT-Aktivitäten, die mit signifikanten Histon-H3- und H4-Deacetylierungsaktivitäten verbunden sind.
Balasubramanyam, K. et al. 2004
Spermidin ist in vitro ein wirksamer Inhibitor von Histonacetyltransferasen.
Spermidin ist ein natürlich vorkommender Autophagie-Induktor als EP300-Inhibitor und erhöht den autophagischen Fluss,
Das Altern ist der dominierende Risikofaktor für die Entwicklung von Herz-Kreislauf-Erkrankungen und führt zu zahlreichen Veränderungen im menschlichen Herzen auf molekularer, struktureller und funktioneller Ebene.
Herzinsuffizienz ist eine altersbedingte Erkrankung, da die Inzidenz mit dem Alter zunimmt. Die autophagische Aktivität des Herzens nimmt während des Alterns ab. Eine kontinuierliche konstitutive Autophagie während des Alterns spielt aber eine entscheidende Rolle bei der Aufrechterhaltung der Herzstruktur und -funktion.
Mikro-RNA lässt neue Herzgefäße sprießen – Schlüsselrolle extrazellulärer Stammzell-Vesikel mit miR-486-5p für das Wachstum neuer Herzkranzgefäße
Gegenwärtige Interventionen können das verletzte Myokard nach einem Infarkt nicht wiederherstellen und erfordern die Entwicklung kardioprotektiver Strategien.
Dagegen ist die Förderung des Blutgefäßwachstums nach einem Myokardinfarkt eine Strategie, um lebensfähiges Myokardgewebe zu erhalten und die Herzfunktion zu verbessern.
Daher besteht ein zunehmendes Interesse an der therapeutischen Verwendung von microRNAs zur Kontrolle der Genexpression durch spezifisches Targeting mit kleinen mRNA Molekülen.
Um Herzkranzgefäße nach einem Herzinfarkt nachwachsen zu lassen, werden die in extrazellulären Vesikel reichlich enthaltene microRNA – miR-486-5p, die aus unter Normoxie oder Hypoxie vorkonditionierten mesenchymalen Stammzellen (MSCs) hergestellt wurde, dem geschädigten Herzen zugeführt.
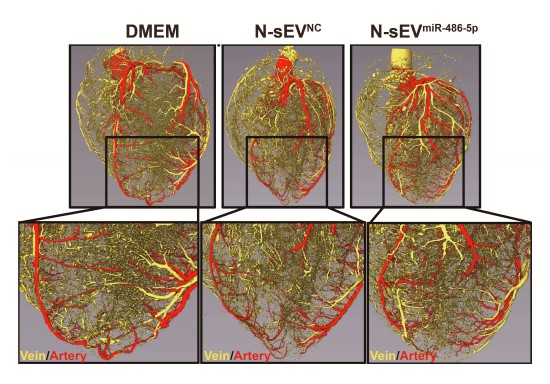
Extrazelluläre Vesikel, die miR-486-5p enthalten, verstärken im Tierversuch bei Mäusen und Primaten, die Angiogenese nach Myocarfinfarkt, indem microRNAs – miR-486-5p -auf fibroblastisches Mmp19 abzielen. Die Mikro-RNA-miR-486- 5p- sorgt dafür, dass das normalerweise vom MmP19-Protein unterdrückte Gefäßwachstum ermöglicht wird und das geschädigte Herz sich regeneriert kann.
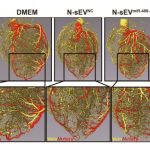
Abb.: Kleine extrazelluläre Vesikel mit viel Mikro-RNA miR486-5p (rechts) regen das Wachstum neuer Blutgefäße in infarktgeschädigten Makaken-Herzen besser an, als Vesikel mit Nährlösung (links) oder funktionsloser RNA (Mitte).
Diese Ergebnisse unterstreichen die Rolle der Mikro-RNA miR486-5p bei der Steigerung der Angiogenese nach Myocardinfarkt.
Referenz: Qingju Li et al: Small extracellular vesicles containing miR-486-5p promote angiogenesis after myocardial infarction in mice and nonhuman primates. Science Translational Medicine 13, 10.03.2021, eabb0202.
Quelle: newsletter epigenetik 34 | april 2021 |
Referenzen auf Anfrage erhältlich.
Ihr
Eduard Rappold
NUGENIS PRODUKTE
Bestellen Sie bequem in unserem Online Shop
Unsere Produkte enthalten pro Kapsel 400mg Ademetionin (S-Adenosyl-L-Methionin) in der biologisch aktiven (S,S) Form in präventiver Dosierung.
Ademetionin ist der singuläre Methylgruppenspender an unser Erbgut (DNA und Histone) und stärkt in diese einmalige Eigenschaft der Genmethylierug die Zellgesundheit.



